:近年来,学科间的交叉会聚越来越明显,科技创新层出不穷。中国科协与生命科学学会联合体、清洁能源学会联合体、信息科技学会联合体、军民融合学会联合体、智能制造学会联合体联合,通过长期的研究,把握世界科技前沿动态,并定期以“中国科协创新智库产品”发布报告。本文主要介绍合成生物学领域近五年国际上的新理论、新原理、新观点、新方法、新、新技术,为科技管理人员了解国内外生命科学的前沿技术及发展趋势提供决策咨询,也为研究与开发人员提供综合的参考信息。
合成生物学的核心思想是在系统生物学的基础上,借鉴工程学思想和现代生物学技术方法来设计和构建新的生物元件、网络和体系,最终人工重构新型的生命体。

CRISPR/Cas9技术:CRISPR/Cas9是一项靶向基因编辑的革新技术,可直接用于基因突变或敲除。相较于传统以基因重组为基础的基因编辑技术,CRISPR/Cas9技术操作简单、成本低、编辑位点精确、脱靶率低,其基因编辑效率超过30%,大大降低了基因编辑的时间成本和经济成本。基因编辑技术是生命科学研究的核心技术之一,通过对基因组序列进行精确地靶向性修改,可以实现人类疾病治疗,以及微生物和动植物的性状改良,在生物医学、农业育种和工业微生物中具有重大的应用前景。
人造生命得以成功,离不开DNA 组装技术的蓬勃发展。近年来,根据型酶切连接、PCR、同源重组、单链退火拼接等原理发展起来的各种 DNA 克隆和组装技术,为合成生物学更加快速而高效地进行段 DNA 的克隆和组装提供技术支持。其后几年,人们致力于更新DNA组装技术,提高重组效率,提高大尺度DNA的组装率。
定向进化指的是在实验室条件下创造突变,并对突变文库筛选压力,从而筛选出具有期望表型的突变体。虽然定向进化是解决合成生物学问题的有力工具,但是其也受到文库大小、筛选效率和进化实验所需时间等因素的。不过,不断出现的新方法已经可以用于缓解定向进化的这些,如:体内定向进化。目前体内定向进化的新技术主要有:

由此可见,体内定向进化策略可以简化定向进化的实验操作,并减少人为因素的干扰,加快了重要功能基因的筛选。
即便拥有如此先进的合成生物学技术,目前人工合成的方法工作量仍然巨大,且成本较高,另外,活细胞内的复杂导致了不可避免的副反应,产物的收率通常很低。无细胞合成生物学(cell-free synthetic biology)由于不需要为细胞提供生长所需的辅助,为解决上述问题提供了有力的支撑。无细胞合成生物学已被研究了数十年,然而直到现在无细胞合成生物学用于工业生产才具有商业可行性。

2014年来自哈佛大学Wyss生物工程研究所、大学等处的研究人员公布了一种基于纸质的非细胞环(纸张基因网络)。为了能现场使用,他们构建了比色法检测,能用于观察,也创造了一种低成本、电子光学界面。这一新技术配备有小和RNA遗传开关驱动,复杂基因环快速构建,程序化体外诊断,以及葡萄糖传感器,特殊埃博拉病毒传感器。这一设计为合成生物学家提供了一种多用途可变系统,将能用于构建低成本,高度可扩展平台,被应用于性快速疾病诊断中。
2016年,美国麻省理工学院(MIT)研究团队提出一个新理论:将不同遗传线包裹在脂质体液滴中,这些脂质体拥有与细胞膜非常相似的脂肪膜,可扮演人工细胞的角色,除了能识别DNA和合成蛋白质外,没有其他任何细胞功能,来它们之间的相互干扰,从而可成更加复杂的遗传线,合成更加复杂的药物。这一工作可以帮助研究人员设计出全新遗传线,并有助于揭开地球生命早期起源之谜。
2016年,来自美国能源部橡树岭国家实验室的研究人员研发出一种无需细胞培养,人工合成蛋白质的新系统。另外大学分校(UCLA)的研究团队利用葡萄糖作为原料,在无需细胞的条件下合成出了相当重要的合成积块(buildingblocks)(乙酰辅酶A,acetyl coenzyme A,acetyl-CoA),既摆脱了活细胞的,又保留了生物合成体系的高效率,为生产塑料、生物燃料和新药铺筑了一条新的道。
合成生物学目前的技术可以实现合成基因、合成通、合成细菌基因组、合成真核染色体,甚至合成整个真核基因组。许多合成生物学的项目具有多种单独的合成目标——“部件”,如启动子、编码区和终止子等。尽管单独的“部件”可以被鉴定,然而预测各个部件如何形成一个整体进行共同运作仍然是一个挑战。
目前国外常用的标准化基因部件的方法,如Golden Gate Assembly,是一个效率高且效果好的组合DNA文库的方法,其整个克隆过程只需要用到1种内切酶,并可以在一个试管中将3~4段不同的基因在一步之内完成拼装。而Golden Gate assembly需要在设计环节进行大规模的计算以产生具有相同粘性末端的标准DNA部件,缺乏预先定义的性内切酶位点等缺点。针对此方法,目前已有的自动提取DNA部件并进行大规模设计的工具有:
劳伦斯伯克利国家实验室的Joanna Chen 于2012年报道了基于网站的生物计算机辅助设计(BioCAD)软件,DeviceEditor。DeviceEditor整合了各种相关资源,通过正确操作软件进行DNA的组装设计,将各种繁杂的任务自动化,可以大大降低研发人员的设计成本,提高设计效率。
J5是一个基于网页的软件工具,由劳伦斯伯克利国家实验室的Nathan J Hillson在2012年开发。它可以自动化地对DNA元件组装方案进行设计。J5大大节省了合成生物学科研人员的研发时间,并降低了劳动强度,减少了研发人员设计的错误率,减少了脱靶的DNA组装产物量,降低了研究成本, 最终使计算机辅助设计多部件组合型DNA构建产物成为可能。
GenomeCarver 在2014年,由英国大学的Emily Scher提出。此软件可以各个DNA部件之间兼容良好,并便捷地衡量各个DNA部件的设计结果。 GenomeCarver填补了合成生物学信息学工具的空白,此工具以及设计构建好的部件库极大地丰富了整个合成生物学领域的研究手段及内容。
为了将设计标准化的生物部件过程进行流程化,约翰霍普金斯大学的kun Yang开发了BiopartsBuilder,它可以从不同的数据库调出序列信息,并出具能够符合设计标准的组合方案。BiopartsBuilder为合成生物领域提供了有用的、新的、集成的、可扩展的功能。
SynBIS是2016年英国帝国理工学院的Dick Kitney等开发的一个基于合成生物学网站的信息系统,其集成了多种BioCAD、各种DNA组装模块以及各种生物部件的特征参数。
编者按:近年来,学科间的交叉会聚越来越明显,科技创新层出不穷。中国科协与生命科学学会联合体、清洁能源学会联合体、信息科技学会联合体、军民融合学会联合体、智能制造学会联合体联合,通过长期的研究,把握世界科技前沿动态,并定期以“中国科协创新智库产品”发布报告。本文主要介绍我国合成生物学领域近五年国内前沿技术、突破性以及取得的国际地位,为科技管理人员了解国内外生命科学的前沿技术及发展趋势提供决策咨询,也为研究与开发人员提供综合的参考信息。
合成生物学的发展很大程度上依赖人们对生命体内在运动规律的掌握,近年来国内的一些研究发现为有目的地生命体提供了理论基础。
2013年,李寅等提出了基于基因组复制工程的连续进化(Genome replication Engineering assisted continuous evolution,GREACE)概念。其原理是通过在微生物细胞中引入一系列经遗传修饰的DNA 聚合酶校正元件使细胞进入一种高突变率的状态,从而在筛选压力条件下加速进化过程。经过一段时间的进化后,将校正元件从细胞中移除,这时细胞能维持稳定的基因型,同时能将这种优势表型遗传下去。这为解决在从头合成基因组中表达异源蛋白时所面对的一些问题提供了有效的解决方法。
2015年,大学自动化系、信息国家实验室生物信息学研究部/合成与系统生物学中心汪小我和谢震研究组了microRNA定量调控规律,发现microRNA的靶位点结合能力对竞争性内源RNA(ceRNA)效应强度影响的函数关系,并阐述了microRNA通和RNAi通竞争效应的不对称性。该研究为合理的设计有效的RNAi实验来减少siRNA的脱靶效应提供了理论基础,为未来用RNAi技术有效设计疾病基因靶向治疗等提供了理论基础。
2016年,中科院深圳先进技术研究院合成生物学工程研究中心研究员刘陈立团队发现细胞通过控制DNA复制的起始来协调细胞个体的大小,二者之间的关系可以用一个简单的数学公式来描述,该公式具有广泛的适用性。基于该定律,结合定量调控特定蛋白表达水平的基因回,人们可以根据实际需求轻松设计并建造出特定长宽的细菌细胞。该研究对实现创造生命这一目标有着积极的意义。定向基因组序列为我们解决其中的某些问题提供了新的思。
2016年,大学医学部周德敏等研究人员利用琥珀密码子(终止密码子)可以识别非天然氨基酸的原理,通过将病毒复制基因的某个或部分编码密码子突变成终止密码子,使其在感染人体细胞后,不能进行完整的蛋白质翻译,从而获得了活病毒疫苗。所获得的病毒疫苗具有安全性和有效性。并且进一步突变三个以上三联码,病毒还由预防性疫苗变为治疗病毒感染的药物,且随着三联码数目的增加而药效增强。这一发现了病毒疫苗研发的,成就了活病毒疫苗的重大突破。
2015年,大学的朱听课题组在Nature Communications在线发表了题为CATCH enables one-step targeted cloning of large gene clusters的论文,他们首次体外应用Cas9系统实现对上百kb的基因组片段的靶向克隆。利用体外转备的sgRNA和表达纯化的Cas9蛋白性酶切目标全基因组,并通过Gibson Assembly原理将长达100 kb的目标片段一步连接到细菌人工染色体上实现性克隆。该技术的开发将极大简化对能够表达具有高附加值生物大的基因簇的克隆步骤,节省时间并降低成本。
2016年,中国科学院上海生命科学研究院的赵国屏课题组利用 CRISPR 相关蛋白 Cpf1开发C-Brick 体外DNA拼接技术。Cpf1 可以由人工设计的 crRNA 性引导切割 DNA并形成 5’突出的粘性末端,通过类似 BioBrick的前后缀设计思,从而实现 DNA 元件的标准化拼接。C-Brick技术从根本上解决了以下两个问题:(1) 所用酶识别位点不能分布的太广泛;(2) 所产生的scar不能太长,不能编码稀有密码子,不能编码极性太强的氨基酸(造成融合蛋白不可溶)。利用C-Brick,赵国屏课题组已成功地拼接了多个荧光基因,进一步促进DNA组装技术的发展。
2016年,中国科学院上海生命科学研究院覃重军团队开发了CasHRA (Cas9-cilitated Homologous Recombination Assembly) 技术。利用酵母原生质体融合,结合 Cas9 在体内切割出待拼接的 DNA 片段,然后利用酵母体内重组系统将片段拼接,最后得到 1.03 M 的MGE-syn1.0 最小大肠杆菌基因组,首次实现兆(M)级别的基因片段的靶向克隆,为合成生物学的进一步发展提供了潜在的研究框架。
这些DNA 组装新技术的不断发展,无论是在尺度上还是效率上,均有了质的飞跃,使得DNA组装技术更简便、高效,且进一步促进了更大更复杂DNA片段组装技术的发展。
由于合成生物学需要基于系统生物学所发现的生命体的运动规律来设计或者一个生命体,因此合成生物学的发展瓶颈主要在于系统生物学。今后生物学家面临的关键问题将不再是如何通过生物学操作来创建新的菌株,而是如何通过模拟分析确定怎样的序列才能使形成的新生命体具有期望的功能。太强调“生物元件”很难找出有普适意义的人工系统,因此我国在开展合成生物学研究时应该走出思维困境,转变科学研究的方式、规范以及战略。
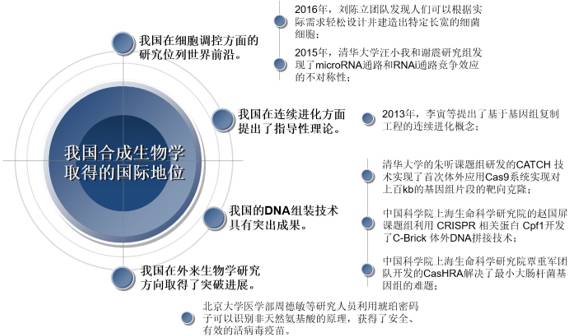
由此可见,我国合成生物学的发展正在逐步追赶发达国家,并形成一套符合我国国情的、具有现实意义导向的,并且能够引领全球合成生物学发展的模式。由于合成生物学应用领域广泛、实用价值高,我国对其发展应保持支持和鼓励的方针,和加强国防、医疗、、材料、经济、能源、食品和农业等领域对合成生物学的学习认识。
推荐:




网友评论 ()条 查看